在jCloud上使用NGS-Analyzer-MINI科研模板
NGS-Analyzer-MINI 基于基因组分析工具NGS Analyzer。NGS 分析仪可以高速分析下一代基因组测序仪产生的输出数据,并可以精确地识别人与人之间的遗传差异或癌细胞的突变。
可以查看下面介绍了解更多信息:
https://github.com/fiber-miniapp/ngsa-mini
创建模板
使用账号登录 jCloud,在左侧的菜单中单击“科研模板”标签,可以看到我们提供的所有科研模板。选择 NGS-Analyzer-MINI。

将鼠标移至模板的图标上,点击“立即创建”,会出现“创建应用”的页面。在这里定义您的模板名称,集群节点数目,每台云主机的登录密码,CPU 和内存的参数,硬盘的类型和大小(实际环境硬盘建议配置至少100G),以及网络带宽和私有网络地址。

在“我的科研应用”标签中,可以看到所创建模板的状态。

等科研模板状态变成“创建完成”。

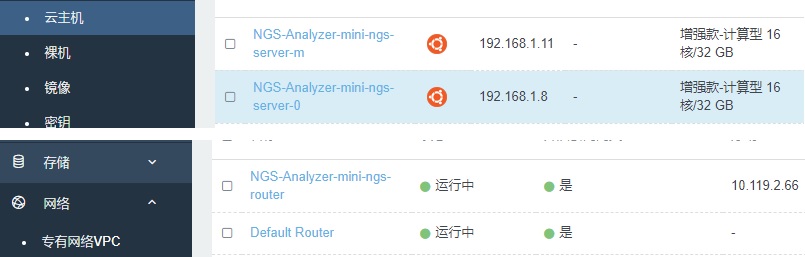
查看云主机列表可以查看新生成的模板主机,查看路由列表可以查看绑定了浮动 IP 的路由器,模板的所有节点可以通过该 IP 与外网进行通信。
使用
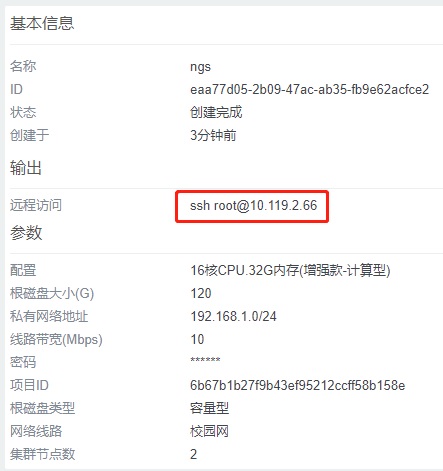
在“科研应用“的”我的科研应用“列表中单击刚创建的应用名称查看详情,在概览页面可以查看到远程访问主节点的方式。
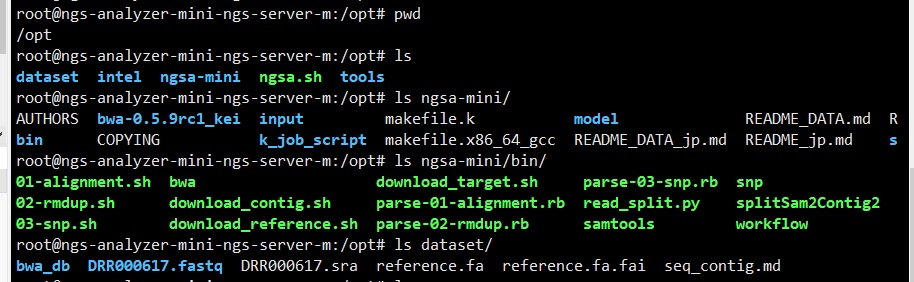
工具安装在 /opt 目录下,包含如下子目录。
先分割测试数据集,参考如下命令(假设使用两个核)
1 | cd /opt/dataset/ |

我们使用以下运行参数进行测试:
1 | cd /opt/dataset |
执行命令后参考的输出结果为:

更多详细参数配置可以参考:
https://github.com/fiber-miniapp/ngsa-mini/blob/master/README.md


 计算
计算  数据库
数据库  存储
存储  网络
网络  安全
安全